데이터를 변수에 저장해줍니다. iris라는 내장데이터를 이용할 것입니다.
data=iris
이후 과정은 번호를 붙여 진행하겠습니다.
1. 데이터 살펴보기
str함수를 이용하여 변수들을 살펴봅시다.
> str(data)
'data.frame': 150 obs. of 5 variables:
$ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
$ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
$ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
$ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
$ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...
종속변수는 Species 입니다. 꽃의 품종을 나타냅니다. 나머지 변수들을 이용해서 Species 를 분류하는 딥러닝 모델을 만들 것입니다.
2. 훈련데이터셋, 테스트데이터셋 나누기
데이터를 훈련데이터셋과 테스트데이터셋으로 나누겠습니다. 전체의 25%를 테스트 데이터셋으로 만들겠습니다. 코드는 아래와 같습니다.
set.seed(999)
id_test=sample(1:nrow(data),nrow(data)*0.25)
data_test=data[id_test,]
data_train=data[setdiff(1:nrow(data),y=id_test),]
3. 모델 만들기
대장암을 예측하는 모델을 만들 것입니다. 딥러닝 알고리즘을 사용합니다. neuralnet 패키지에 있는 함수를 사용합니다. neuralnet 함수이고 hidden 옵션을 이용해서 뉴런 수와 레이어 수를 결정합니다. c(3,2) 는 두 층으로 되어 있고 1층 뉴런이 5개, 2층 뉴런이 2개라는 의미입니다.
#4.모델 만들기
library(neuralnet)
model_nn = neuralnet(
Species ~ Sepal.Length+Sepal.Width+Petal.Length+Petal.Width,
data = data_train,
hidden = c(5,2)
)
모델 생성 결과는 아래와 같습니다.
> plot(model_nn)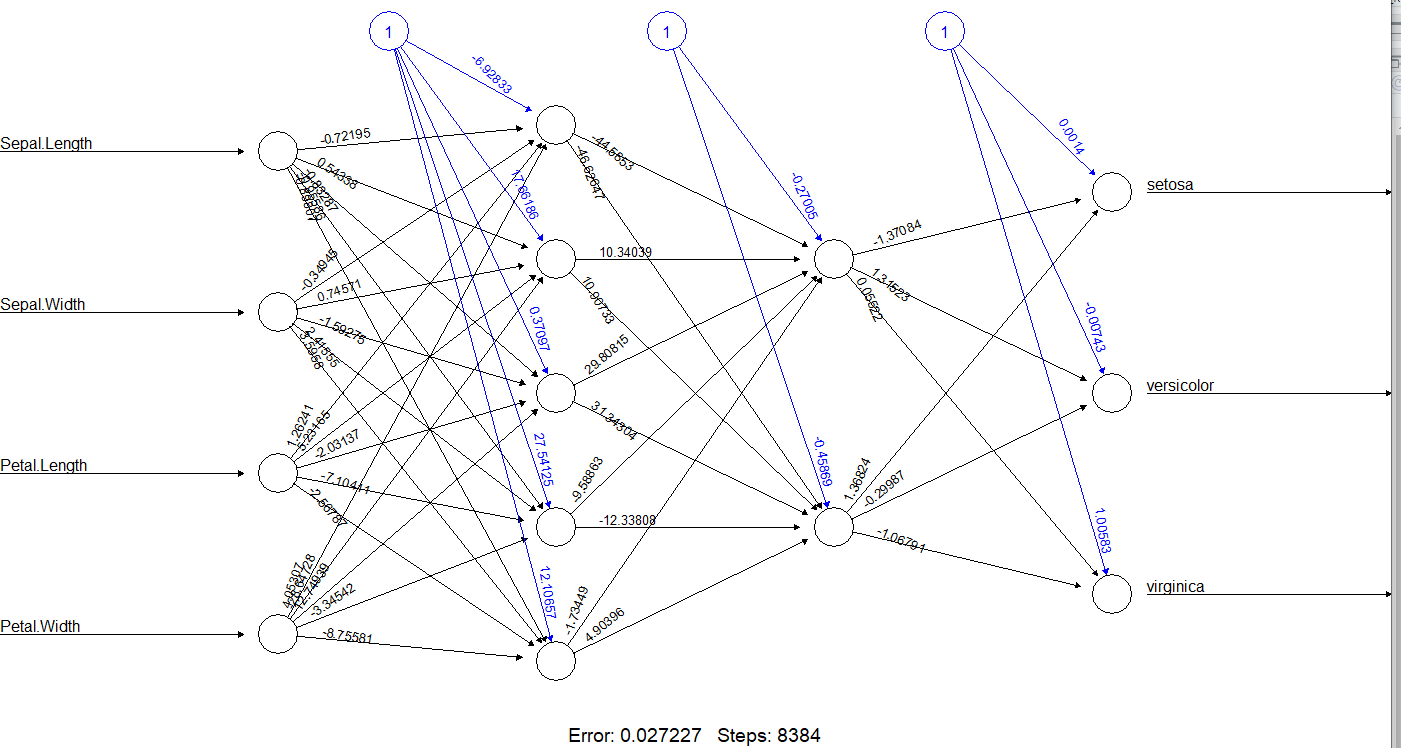
4. 모델 평가
predict 함수를 이용하여 테스트셋의 결과변수를 구해줍니다. type 을 response 로 설정하면 확률이 반환됩니다. 해당 확률이 0.5보다 높은 경우를 1로, 낮은 경우를 0으로 반환한 뒤, test 데이터의 status 와 비교합니다. 각 종의 예측정확도를 각각 평가해야 합니다.
#5.모델 평가하기
pred = predict(model_nn, data_test, type = 'response')
#setosa 예측 정확도
mean(ifelse(pred[,1] > 0.5, 1, 0) == (data_test$Species=='setosa'))
#versicolor 예측 정확도
mean(ifelse(pred[,2] > 0.5, 1, 0) == (data_test$Species=='versicolor'))
#virginica 예측 정확도
mean(ifelse(pred[,3] > 0.5, 1, 0) == (data_test$Species=='virginica'))
test 데이터에 대해 ROC 곡선도 그려봅시다.
par(mfrow=c(2,2))
#roc curve
library(Epi)
ROC(test = pred[,1],
stat = (data_test$Species=='setosa'),
plot = "ROC")
ROC(test = pred[,2],
stat = (data_test$Species=='versicolor'),
plot = "ROC")
ROC(test = pred[,3],
stat = (data_test$Species=='virginica'),
plot = "ROC")
코드 모아보기
library(survival)
#1.데이터
data = colon
#2.결측치확인 및 제거
#결측치 위치 확인 함수
where.na.df = function(df) {
res = data.frame(row = NA, col = NA)
for (i in 1:dim(df)[1]) {
for (j in 1:dim(df)[2]) {
if (is.na(df[i, j])) {
res = rbind(res, c(i, j))
}
}
}
res = res[-1, ]
rownames(res) = NULL
return(res)
}
where.na.df(data) #결측치 위치확인
data = na.omit(data) #결측치 제거
#2.훈련데이터셋, 테스트데이터셋 나누기
set.seed(999)
id_test = sample(1:nrow(data), nrow(data) * 0.25)
data_test = data[id_test, ]
data_train = data[setdiff(1:nrow(data), y = id_test), ]
#3.모델 만들기
model_svm = svm(
status ~ rx + sex + age + obstruct + perfor + adhere + nodes + differ +
extent + surg,
data = data_train,
kernel = "radial",
probability = TRUE
)
#4.중요도 출력
library(caret)
varImp(model, scale = FALSE)
#6.모델 평가하기
pred = predict(model, data_test, type = 'response')
mean(ifelse(pred > 0.5, 1, 0) == data_test$status)
#roc curve
library(Epi)
ROC(test = pred,
stat = data_test$status,
plot = "ROC")
댓글